基于TransH的医疗知识图谱问答系统
项目总结报告
简介
概述
本项目结合Django框架搭建一个以疾病为中心的一定规模医药领域知识图谱,并以该知识图谱完成自动问答与分析服务。以医疗知识图谱为基础,以疾病为核心,本项目构建起一个包含9259个实体和16732个实体关系的知识图谱。 本项目将包括以下部分的内容并会在第二部分详细阐述:
(1)知识抽取:将网页寻医问药网作为来源,使用爬虫技术爬取相关医药领域的知识;抽取 node 的模块,关系分类的模块。我们使用 MongoDB 和 python 的 pymongo 库来对数据进行暂时的存储和爬取操作,将最终爬取的数据进行处理并导出为 json 格式文件。
(2)知识存储:采用 Neo4j 数据库来对获取数据进行存储,在 python 文件中通过 py2neo 连接数据库,将获取的 json 格式文件的数据导入 Neo4j 数据库中。
(3)向量表示:实现 TransH 算法,将实体和关系转化为向量。
(4)知识推理:实现基于知识库的查询检索和问答模块。
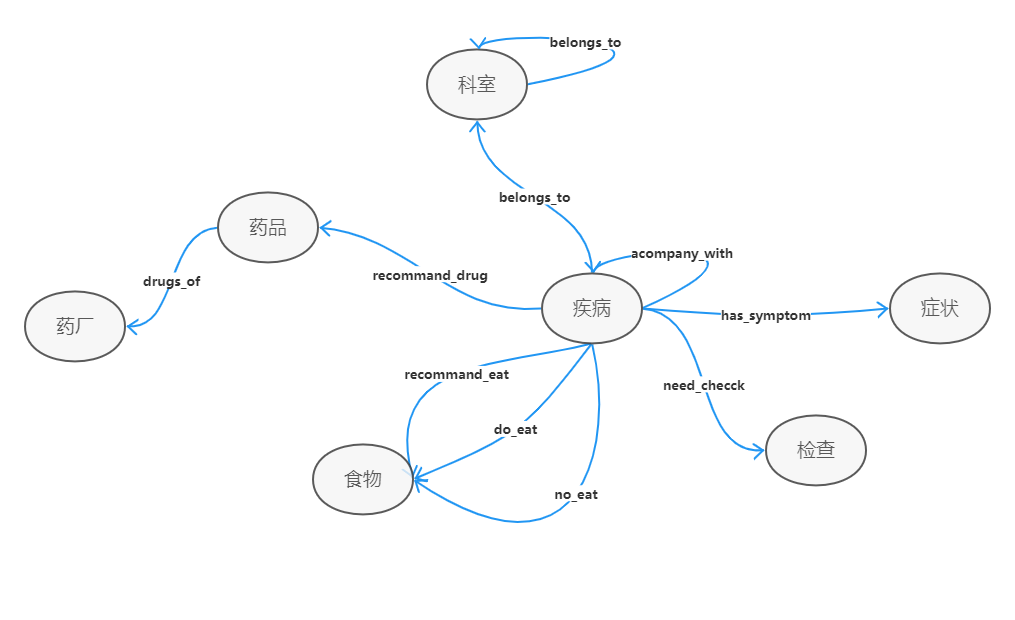
本系统的知识图谱Schema如下:
代码文件说明
-
爬虫对应代码文件:
Spider.py
-
数据构建对应代码文件:
Data2Mongo.py
Cut_Words.py
-
知识存储对应代码文件:
Data2Neo4j.py
-
向量表示对应代码文件:
TransH.py
-
查询测试对应代码文件:
Test.py。
-
问答系统对应代码文件:
Q_Class.py
Q_Parser.py
Match.py
A_byMatch.py
A_byTransH.py
参考资料
功能实现
知识抽取
数据爬虫
爬虫思路:解析网页→提取链接→发送请求→接收响应→提取资源→存储资源。
1.我们从网页寻医问药网获取疾病信息,该网站网址组织的方式为root网址+信息+疾病编号。我们通过循环确定每个疾病信息的url进行信息访问,如代码1。
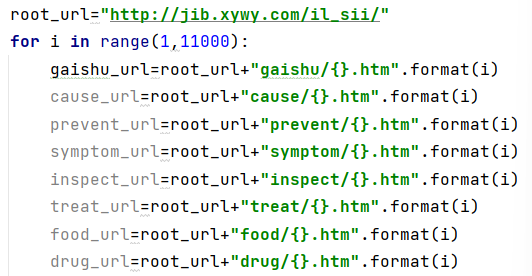
2.根据寻医问药网对疾病信息的组织方式,对所要爬取的网页利用 python requests 库中的 get 方式进行请求、以获取其相应返回的结果。
3.然后再使用 xpath 和 etree 等技术方法,对响应内容解析,搜索HTML文档内提取想要的内容资源,并做一些去除空格、tab键和换行符的处理。
4.最后,将提取出来的资源保存起来。我们将最终得到的每个疾病的信息以字典的形式存储在 MongoDB 数据库中。
爬虫对应代码文件:Spider.py。为处理不同网页下的信息,我们在类内组织了7个函数gai_spider(self, url)、cas_pre_spider(self, url)、symptom_spider(self, url)、inspect_spider(self, url)、treat_spider(self, url)、drug_spider(self, url)、food_spider(self, url)、inspect_crawl(self)对应处理疾病的概述、病因、症状、检查、治疗、药物、饮食保健等信息。
MongoDB数据库中存储每个病症的字典的结构如下:
data={
'gaishu':{'category','name','desc','attributes'},
'symptom':{'symptoms','symptoms_detail'},
'food':{'good','bad','recommand'},
'drug','cause','prevent','inspect','treat'
}
数据构建
数据处理思路:对爬虫得到的存储于 MongoDB 的数据进行进一步处理和调整,将疾病的具体属性下的信息梳理到更规范的字典中,其结构如下图2。此时字典中对应的键值是初始值,根据爬虫得到的 data 信息中获取并处理后填充初始值。最终处理后的信息将再次放到 MongDB 数据库中。
data_modify = {
#从data['gaishu']中获取
'名称': 'name',
'所属类别': 'category',
'简介': 'desc',
'医保疾病' : 'yibao_status',
"患病比例" : "get_prob",
"易感人群" : "easy_get",
"传染方式" : "get_way",
"就诊科室" : "cure_department",
"治疗方式" : "cure_way",
"治疗周期" : "cure_lasttime",
"治愈率" : "cured_prob",
'常用药品' : 'common_drug',
'治疗费用': 'cost_money',
'并发症': 'acompany'
#从data['drug']中获取
'药品明细': 'drug_detail',
'药品推荐': 'recommand_drug',
#从data['food']中获取
'推荐': 'recommand_eat',
'忌食': 'not_eat',
'宜食': 'do_eat',''
'预防措施': 'prevent',#从data['prevent']中获取
'成因': 'cause',#从data['cause']中获取
'症状': 'symptom', #从data['symptom']中获取
'检查': 'check',#从data['food']中获取
}
数据构建对应代码文件:Data2Mongo.py,Cut_Words.py。
在对数据进行处理中,使用双向最大向前匹配进行分词,我们采用的方法的思路是是将正向最大匹配法得到的分词结果和逆向最大匹配法的到的结果进行比较,从而决定正确的分词方法。如果正反向分词结果词数不同,则取分词数量较少的那个。如果分词结果词数相同 :若分词结果相同,就说明没有歧义,可返回任意一个;若分词结果不同,返回其中单字较少的那个。
知识抽取结果:最终处理好的数据,我们直接从 MongoDB 中导出,存储到文件 medical.json 中,以方便后续的处理。
知识存储
采用图形数据库 neo4j 数据库来对获取的数据进行存储,在 python 文件中通过 py2neo 连接数据库将获取数据导入。
知识图谱构建:
我们爬取实体有 9259 个,关系有 16732 个。
实体分为检查指标如血压、血脂等,疾病名称,疾病症状,疾病科室,药厂,药品,食物,并在neo4j中建立相应节点。
关系分为 acompany_with:并发症,belongs to:疾病属于哪个科室,do_eat:宜吃,drugs_of:药厂产药,has_symptom:症状,no_eat:忌食, recommend_drug:好评药品 ,recommend_eat:建议食谱。
| start_node | end_node | edge | relation_type | relation_name |
|---|---|---|---|---|
| 疾病 | 食物 | rels_recommandeat | recommand_eat | 推荐食谱 |
| 疾病 | 食物 | rels_noteat | no_eat | 忌吃 |
| 疾病 | 食物 | rels_doeat | do_eat | 宜吃 |
| 科室 | 科室 | rels_department | belongs_to | 属于 |
| 药厂 | 药品 | rels_drug_producer | drugs_of | 生产药品 |
| 疾病 | 药品 | rels_recommanddrug | recommand_drug | 好评药品 |
| 疾病 | 检查 | rels_check | need_check | 诊断检查 |
| 疾病 | 症状 | rels_symptom | has_symptom | 症状 |
| 疾病 | 疾病 | rels_acompany | acompany_with | 并发症 |
| 疾病 | 科室 | rels_category | belongs_to | 所属科室 |
知识存储对应代码文件:Data2Neo4j.py。
向量表示
向量表示思路:使用的算法为 TransH 算法。
训练实现:
将neo4j数据库中的关系和实体导入到列表中,整理为三元组,三元组中存放的为实体和关系在neo4j中的id,将实体和关系表示为向量方便后面做推理。我们有9000个实体,16000个关系,我们构建两个矩阵,第一个矩阵的维数为 200*9000代表实体,另一个矩阵的维数为200*16000代表关系。这样实体和关系就被嵌入了200维的向量。在最开始训练时,实体和关系的普通向量和映射向量中的值是随机初始化的,初始化时加上限制他们均匀分布在 (-\frac{6}{k},\frac{6}{k}) 之间,其中 k就是嵌入维数,k=200。我们用字典的存储方式实现 neo4j 的id映射到随机初始化的向量上,然后在训练中调整向量表示。
训练轮数 epoch 设为100。每一轮采样 batchsize 数量的三元组,将部分三元组通过替换头尾节点进行负采样。使用 Pytorch 中的优化器,随机梯度的方法根据下式表示的 loss 函数对向量进行更新。
向量表示对应代码文件:TransH.py。
知识推理
查询
查询匹配思路:对于所有的三元组,分别对每一个三元组进行如下操作:将三元组中的头节点作为缺失的部分,带入相应集合中的所有实体,根据向量表示TransH算法中的结果,计算出距离,并对集合所有实体的距离进行排序。由此可计算出前十个命中正确头节点的概率以及正确头节点的平均排名。
查询对应代码文件:Test.py。
问答系统
将问题带入提前准备好的知识库寻求答案的一种基于知识库的问答系统。该问答系统可以解析输入的自然语言问句,主要运用对象正则表达式匹配得到结果,然后利用对应的查询语句,请求后台基于 Neo4j 知识谱图数据库的服务,最终得到我们想要的结果。
问答系统思路:对用户提出的问题问题先进行分词,根据对应各种关系的字典,匹配出用户所问的问题,然后可以进行1)构造SQL语句查询,2)或者根据用户的问题,结合TransH算法计算向量匹配。最终罗列出排名靠前的几项匹配结果供用户参考
问答系统对应代码文件:
1.Q_Class.py
该文件用于将用户问题的问句类型进行分类。
| 函数名称 | 输入 | 输出 | 功能 |
|---|---|---|---|
| QuestionClassifier.init(self) | 无 | 无 | 初始化函数,读取特征词字典文件,构建特征词抽取AC自动机,构建特征词种类字典,构建问句疑问词字典,打印提示信息“model init finished ......” |
| QuestionClassifier.classify(self,question) | 问句字符串question | 字典data,‘args’项为抽取出的特征词及种类,‘question_types’项为问句类型 | 分类主函数,抽取问句特征词及种类存入data字典args项,根据问句中包含的问句疑问词和特征词种类判断得出问句类型,存入data字典question_types项,最后返回data字典。 |
| QuestionClassifier.build_wdtype_dict(self) | 无 | 字典wd_dict,由特征词字典中所有特征词和其对应种类组成,索引为特征词,值为对应种类 | 功能函数,根据各类特征词字典得出特征词总字典中的特征词对应种类,存入wd_dict字典中。 |
| QuestionClassifier.build_actree(self,wordlist) | 提取词列表wordlist | 构建的特征词提取AC自动机actree | 功能函数,调用ahocorasick包函数Automation()初始化actree,调用actree自带函数add_word遍历提取词列表中索引和词加入actree中,最后调用actree自带函数make_automation()完成词的加入。 |
| QuestionClassifier.check_medical(self,question) | 问句字符串question | 字典final_dict,由问句中提取的特征词及其种类组成,键为特征词,值为其种类 | 功能函数,调用构建好的AC自动机region_tree提取问句中特征词,对提取的特征词进行去重,再从特征词及种类字典wd_dict中得出提取出的特征词种类,和特征词一起存入final_dict字典中,键为特征词,值为其种类。 |
| QuestionClassifier.check_words(self,wds,sent) | 查找字列表wds,查找环境sent | BOOL值True or False,即环境中是否含有查找词 | 功能函数,遍历字列表判断是否在环境中,任一个在则返回True,遍历结束返回False。 |
| 主程序(仅测试用) | / | / | QuestionClassifier类实例化,死循环输出提示信息“input a question:”,读取问句,对问句调用classify函数得到data字典并输出。 |
2.Q_Parser.py
该文件用于解析问句并生成sql查询。
| 函数名称 | 输入 | 输出 | 功能 |
|---|---|---|---|
| QuestionParser.build_entitydict(self,args) | 字典args,键为特征词,值为其种类 | 字典entity_dict,键为特征词种类,值为该种类的所有特征词 | 功能函数,遍历字典args,设置种类为entity_dict字典键,将特征词根据其种类存入对应键的值中。 |
| QuestionParser.parser_main(self,res_classify) | 字典res_classify,‘args’项为抽取出的特征词及种类,‘question_types’项为问句类型 | 查询sql语句列表sqls,每一项为一个字典,字典‘question_type’项为问句类型,‘sql’为数据库中查询的具体sql语句 | 解析主函数,遍历问句类型的每一个值,根据问句类型向类QuestionParser功能函数sql_transfer传入问句类型和问句提供的对应种类的特征词,问句类型存入字典sql_question_type项中,功能函数sql_transfer输出的数据sql查询语句存入字典sql_args项中,最后将字典sql_加入到列表sqls,遍历结束返回sqls列表。 |
| QuestionParser.sql_transfer(self,question_type,entities) | 问句类型question_type,问句中实体列表entities | 转换后的数据库中查询的具体sql语句sql | 功能函数,根据问句类型,利用问句提供的对应类型的实体写出具体的数据库sql查询语句。 |
| 主程序(仅测试用) | / | / | QuestionParser类实例化。 |
3.Match.py
该文件用于答案的查询。
| 函数名称 | 输入 | 输出 | 功能 |
|---|---|---|---|
| AnswerSearcher.init(self) | 无 | 无 | 初始化函数,连接数据库,并限制返回最大答案数20。 |
| AnswerSearcher.search_main(self,sqls) | 具体sql查询语句列表sqls | 具体回答字符串列表final_answers,实体关系三元组列表web_answers | 查询主函数,遍历sqls列表每一项,根据项中提供的问句类型和sql查询语句,调用数据库进行查询得到返回值,根据返回值得到实体关系三元组存入web_answers列表中,再进行具体问句回答形式优化,存入final_answers列表中并返回列表。 |
| AnswerSearcher.prettify(self,question_type,answers) | 问句类型question_type,数据库查询返回值列表answers,每一项为字典,字典项‘m.name’存储头节点名称,‘r.name’存储关系名称,‘n.name’存储尾节点名称 | 形式优化后的回答字符串final_answer | 功能函数,根据具体问句类型优化回答形式,形成完整句子final_answer字符串。 |
| 主程序(仅测试用) | / | / | AnswerSearcher类实例化 |
4.A_byMatch.py
该文件调用其他三个代码文件实现问答程序。
| 函数名称 | 输入 | 输出 | 功能 |
|---|---|---|---|
| ChatBotGraph.init(self) | 无 | 无 | 初始化函数,进行QuestionClassifier类、QuestionParser类和AnswerSearcher类的实例化。 |
| ChatBotGraph.chat_main(self,sent) | 问句字符串sent | 回答字符串answer,或真实回答和实体关系三元组列表 | 问答主函数,首先判断问句是否为“退出”,是则返回‘exit’表示退出,否则对问句进行提取特征词及种类和问句类型,再根据此结果解析得到sql查询语句,最后在数据库中进行查询得到最终回答,回答为空则返回‘未找到相关信息。’的提示信息,否则返回真实回答和实体关系三元组列表。 |
| 主程序 | / | / | ChatBotGraph类实例化,调用问答主函数,并返回真实回答和实体关系三元组列表。 |
实验结果
查询
测试集划分及结果:
为了测试训练效果,我们将样本划分为训练集与测试集,具体操作如下:在所有三元组中选择
\frac{4}{5}的三元组作为训练集,选择\frac{1}{5}的三元组为测试集进行5折交叉验证。训练后,对于剩余的三元组,我们将其头结点和为节点进行替换,替换过程为遍历实体集并依次计算head,relation,tail的距离来进行排名。根据测试集正确结果所在的距离排名来计算 hit2,hit10 。(hit \space i)代表正确结果在前i名的概率。
我们通过增大嵌入维数、提高epochs等来获得较高的正确率。最终,我们在训练集 loss 为 0.003,测试集为
hit2 为0.985, hit10 为1 ,取得了不错的效果。
测试对应代码文件:Test.py
问答系统
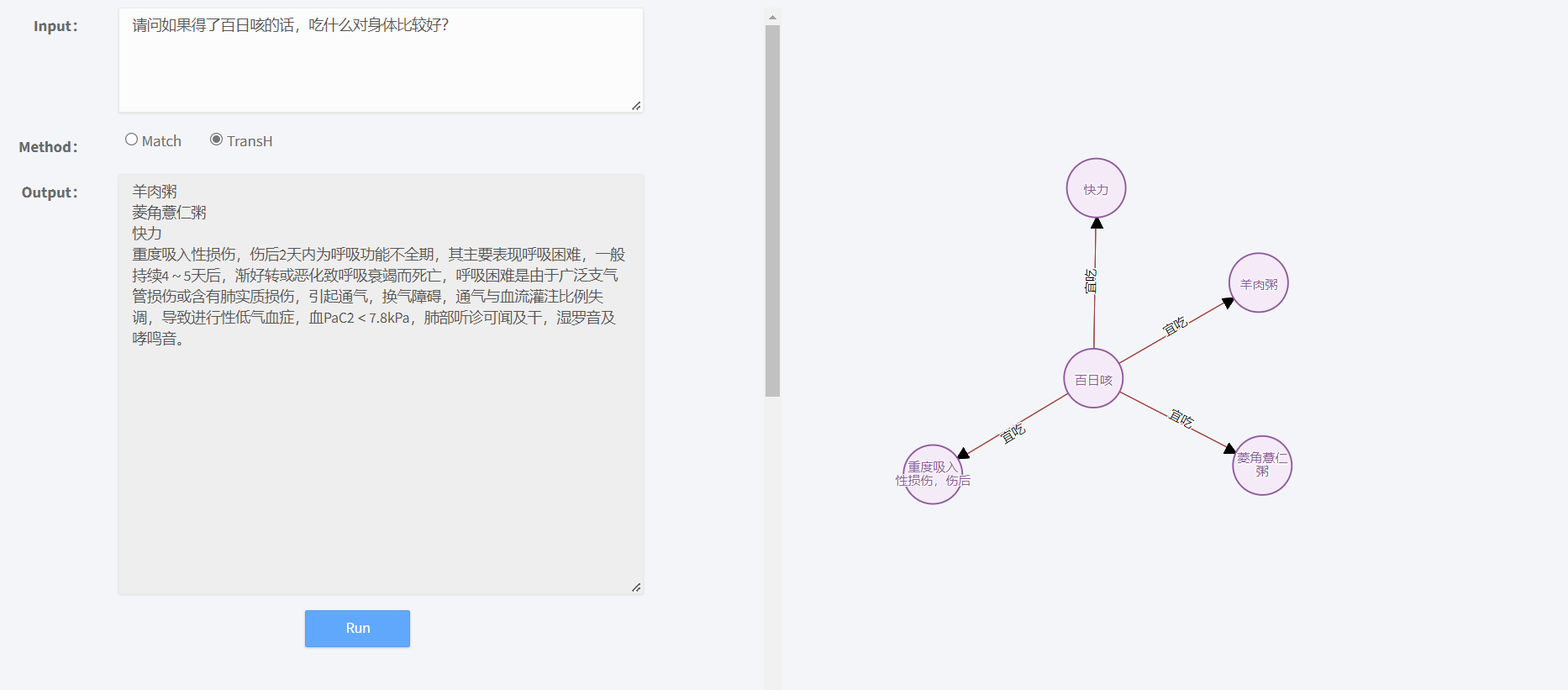
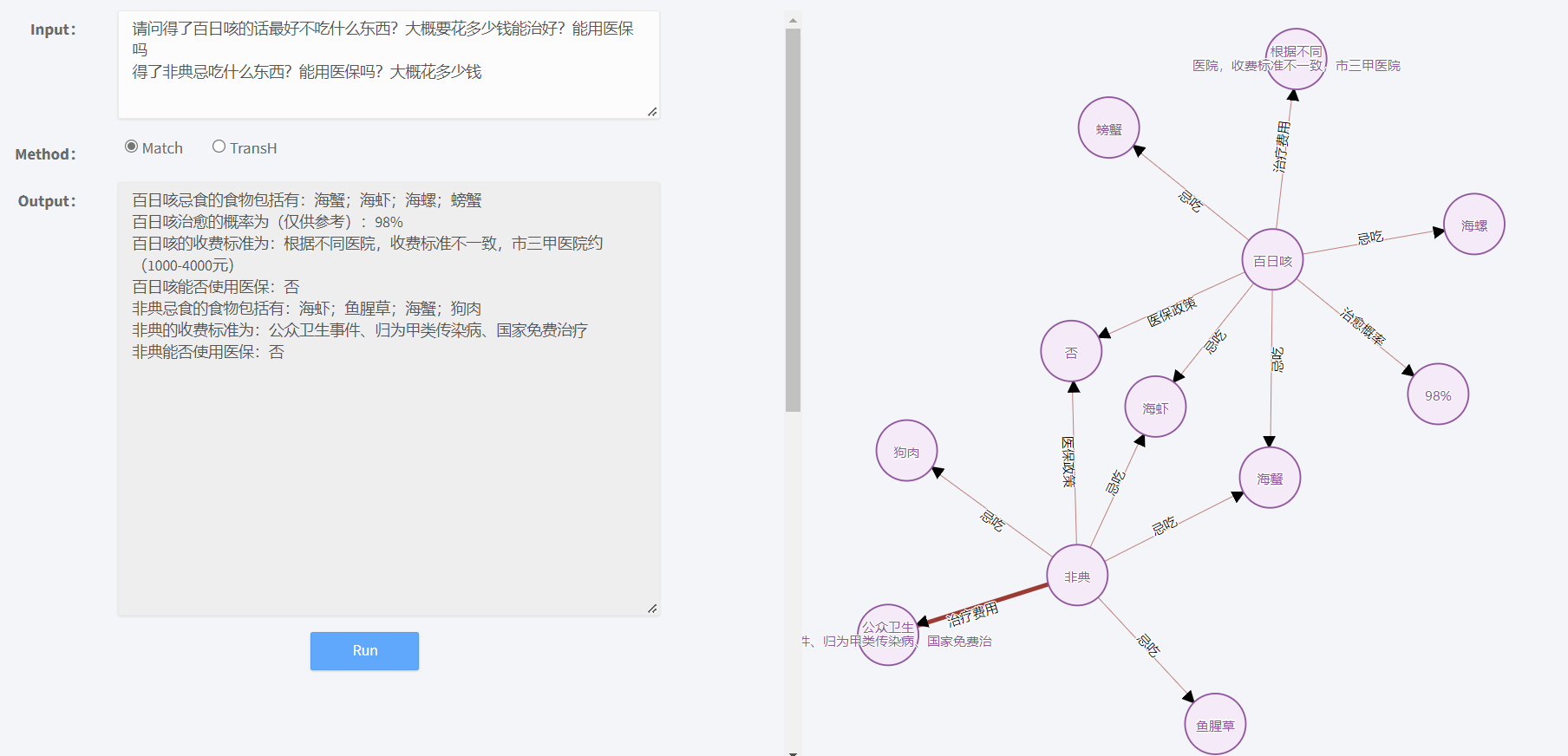
用户可以选择两种方法Match和TransH来查找问题相关的信息,除了文字类型的信息,右图还会以图的形式给出知识图谱里相关的结构关系。
总结
本项目搭建一个以疾病为中心的一定规模医药领域知识图谱,并以该知识图谱完成自动问答与分析服务。以医疗知识图谱为基础,以疾病为核心,基于TransH、etree、pytorch等技术上,具有集成性,先进性,统一性,完整性和开发性。我们通过知识抽取、知识存储、向量表示和知识推理的步骤来实现整个系统的功能。未来我们希望可以在现有的基础上扩充知识图谱的内容,增加更多的关系;提高对问答系统的准确度和智能问答的交互体验。
